Evolutionaire conflicten voorspeld
Evolutie lijkt een onvoorspelbaar proces. Maar het voorspellen van de grenzen van evolutie blijkt wel mogelijk. Dit tonen onderzoekers van AMOLF en het Franse ESPCI aan door middel van hun wiskundige methode gevolgd door experimenten met bacteriën. Op 24 juni publiceerden ze hun resultaten in het vaktijdschrift Cell Systems.

Cellen maken gebruik van nauwe samenwerking tussen eiwitten. Dit soort regulatienetwerken stelt cellen in staat om hun omgeving te interpreteren, en zo het juiste gen ‘aan’ te zetten; bijvoorbeeld om het enzym tot expressie te brengen dat antibiotica kan afbreken. Voor bacteriële cellen is dit essentieel om te kunnen overleven, en hun regulatienetwerken staan daarom continue onder evolutionaire selectie. Maar hoe zij uiteindelijk evolueren is moeilijk te voorspellen.
De onderzoekers kozen voor de volgende aanpak. Ze maakten eerst een nieuw genetisch netwerk in de bacterie E. coli dat twee omgevingsmoleculen (A en B) in staat stelt om de expressie van een fluorescent eiwit te beïnvloeden. In plaats van de evolutie van dit netwerk te voorspellen door naar willekeurige mutaties te kijken, richtten de onderzoekers zich op alle mogelijke toekomstige functies van het netwerk, en de grenzen hiervan. Een van de vragen die ze zo konden stellen was: ‘Kunnen we voorspellen of evolutie ervoor kan zorgen dat de cellen alleen fluorescent worden als molecuul A aanwezig is, en molecuul B niet’?
De onderzoekers ontwikkelden hiervoor een nieuwe wiskundige methode. Met een grafische aanpak bleek het eenvoudig om de evolutiegrenzen van het genetische netwerk te bepalen. Deze analyse gaf aan dat het hierboven beschreven evolutionaire doel niet eenvoudig haalbaar is. Alleen met een verregaande evolutionaire aanpassing, bijvoorbeeld in de detectie van molecuul A zelf, zou dit kunnen.
Om deze wiskundige voorspelling te testen, lieten de onderzoekers de bacteriën op een gecontroleerde manier evolueren. Ze lieten eerst een grote populatie van bacteriën willekeurig muteren, en groeiden ze daarna samen in een erlenmeyer zonder A en B, en daarna met A maar zonder B, etc. Ze richtten daarbij de experimenten zo in, dat er een competitie tussen verschillende mutanten ontstond, waarin de mutant die zijn taak het best uitvoert zich het meest vermenigvuldigt. De resultaten lieten precies zien wat voorspeld was: in het begin van de evolutie kwamen de bacteriën dichtbij de goede oplossing, maar niet helemaal. Ze reageerden ofwel goed op A, ofwel goed op B, in plaats van goed te reageren op beide signalen. Er ontstond een soort conflict tussen twee evolutionaire doelen. Na langere evolutie bleek het wel mogelijk het conflict op te lossen – en een genetische analyse liet toen inderdaad een verregaande innovatie van een van de eiwitten zien.
Het kunnen voorspellen is een heilige graal binnen de evolutie. Voorspellen is in sommige disciplines zoals engineering of natuurkunde heel normaal, maar binnen de biologie en zelfs evolutie is dit veel lastiger. Er zijn nu weer veel discussies over de mogelijkheden of onmogelijkheden van voorspellen, en wat hier nodig voor zou zijn. Praktische concepten en experimenten zoals deze geven hierin een begin van een antwoord.
Schematische illustratie van het netwerk in E. coli (links), en het resultaat van de evolutie experimenten en voorspellingen (rechts). Het netwerk detecteert twee moleculen Dox en IPTG, en laat als response een set genen (lacZ, cmR, en sacB) tot expressie komen. Het netwerk wordt gemuteerd door middel van een PCR-reactie, en de set genen zorgen voor de natuurlijke selectie. Rechts zijn drie assen te zien die de groeisnelheid in drie verschillende omgevingen uitdrukken. De blauwe en groene lijnen geven de voorspelde evolutie-grenzen. De punten zijn de geevolueerde E. coli stammen, beginnend bij het groene punt en eindigend bij het rode. De tussenliggende zwarte punten volgen de voorspelde grenzen.
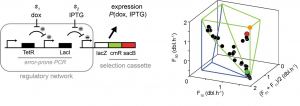
Schematische illustratie van het netwerk in E. coli (links), en het resultaat van de evolutie experimenten en voorspellingen (rechts). Het netwerk detecteert twee moleculen Dox en IPTG, en laat als response een set genen (lacZ, cmR, en sacB) tot expressie komen. Het netwerk wordt gemuteerd door middel van een PCR-reactie, en de set genen zorgen voor de natuurlijke selectie. Rechts zijn drie assen te zien die de groeisnelheid in drie verschillende omgevingen uitdrukken. De blauwe en groene lijnen geven de voorspelde evolutie-grenzen. De punten zijn de geevolueerde E. coli stammen, beginnend bij het groene punt en eindigend bij het rode. De tussenliggende zwarte punten volgen de voorspelde grenzen.
Referentie
Manjunatha Kogenaru, Philippe Nghe, Frank J. Poelwijk, Sander J. Tans, Predicting Evolutionary constraints by identifying conflicting demands in regulatory networks, Cell Systems 10, 1-9, June 24 (2020).


